前回記事では、DNAがどのように複製されるのかを見てきました。
DNAはタンパク質の合成に必要な遺伝情報を含んでいますが、細胞の中ではこの遺伝情報をもとに日々どのようにしてタンパク質が作られているのでしょうか。
今回は、その基本となる「転写」と「翻訳」という2つのプロセスを解説します。
目次
セントラルドグマ
生物の細胞内では、DNAに書き込まれた遺伝情報に基づいて、まずmRNAという物質が合成され(転写)、続いてこのmRNAに基づいてタンパク質が合成されます(翻訳)。
この一連の流れはセントラルドグマと呼ばれ、分子生物学の基本概念となっています。
ざっくりとしたイメージをつかむために例えて言うなら、
DNAは、タンパク質という料理を作るためのレシピが書かれた部厚いレシピ本のような役割です。そこには様々なメニューの作り方が書かれています。
転写は、分厚いレシピ本の中から欲しいメニューを作るために、必要なレシピが書かれた部分を書き写す工程にあたります。
翻訳は、そのレシピメモをもとに、材料であるアミノ酸を運んできて、タンパク質という料理を作る工程にあたります。
前回紹介したDNAの複製は、おおもとのレシピ本を2つ作るというイメージでしたが、
転写の場合はおおもとのレシピ本は増やすことなく、一時的なメモを作るイメージなのです。
メモにあたるmRNAは、一時的なものであるため、役割が済むとすみやかに分解されます。
転写のしくみの前に ― 鋳型鎖とRNAの特徴
具体的な転写の流れを説明する前に、鋳型鎖と非鋳型鎖、DNAとRNAの違いについて先に確認しておきましょう。
鋳型鎖と非鋳型鎖
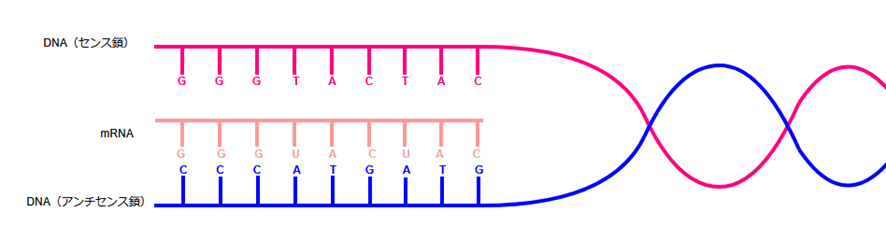
DNAは二重らせん構造で、2本の鎖が向かい合って並んでいますが、転写のときに使われるのはこのうちの片方だけです。
この転写に使われる側の鎖を「鋳型鎖」または「アンチセンス鎖」と呼びます。
鋳型鎖は、RNAポリメラーゼという酵素がmRNAを作るときの元になります。この鋳型鎖に合わせて、mRNAが1本だけ合成されます。
一方、転写に使われない残りの一方は「非鋳型鎖」または「センス鎖」といいます。
mRNAは鋳型鎖に相補的な塩基配列を持つため、mRNAの配列はセンス鎖とほぼ同じ並びになります(後述のようにTがUに置き換わる点のみ異なります)。
このように、タンパク質はmRNAの配列をもとに合成されるため、mRNAと同じ意味を持つこのDNA鎖を「センス(sense)鎖」と呼びます。
DNAとRNAの違い
RNAポリメラーゼは、DNAの二重らせんをほどきながら、元のDNAの塩基と相補的な塩基を細胞質中から取り込んでRNA鎖を伸ばしていきます。
この基本的な流れはDNAポリメラーゼがDNAを複製するプロセスと似ていますが、RNAはDNAとは構造的な違いがあります。
以下では、DNAとRNAの違いについて具体的に見ていきましょう。
ヌクレオチドの2つの違い
DNAはデオキシリボヌクレオチドというユニットが連なって構成されていますが、RNAの場合はリボヌクレオチドで構成されているという点が異なります。
デオキシリボヌクレオチドとリボヌクレオチドには、2点の違いがあります。
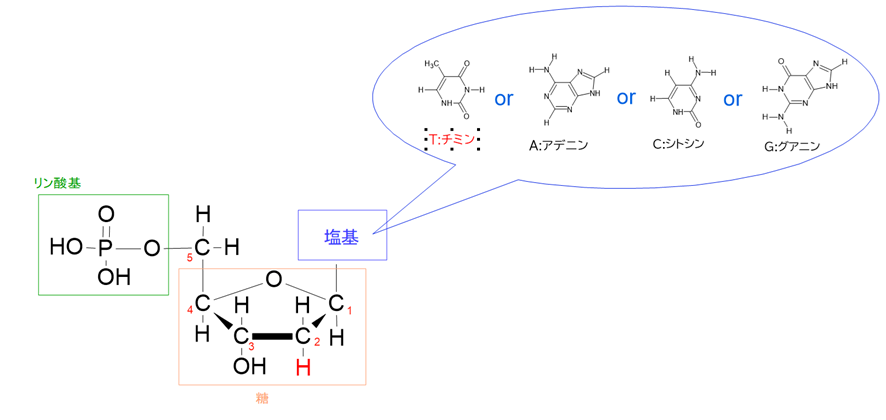
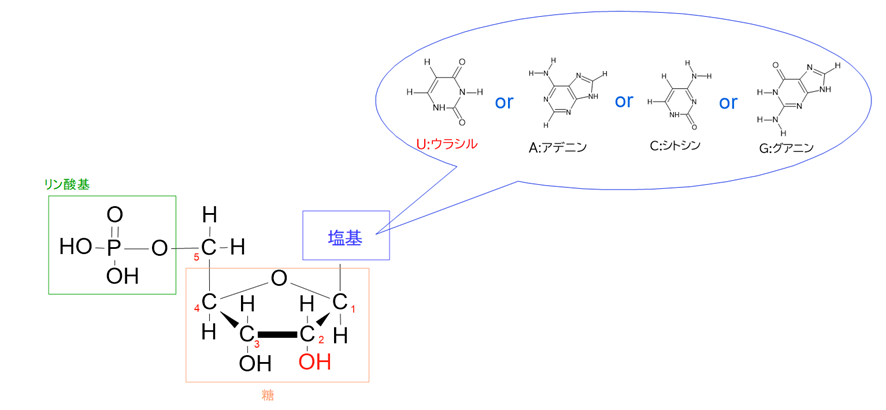
1つ目は、糖の構造の違いです。
DNAに含まれる糖(デオキシリボース)では、炭素番号2番目(2’位)の位置に水素原子がついています。
一方、RNAに含まれる糖(リボース)では、同じ位置にヒドロキシ基(OH)がついています。
このように、RNAの糖には酸素を含むOH基があるのに対し、DNAの糖は酸素が「欠けている」ため、「デオキシ(脱酸素)」リボースと呼ばれるのです。
2つ目は、塩基の種類です。
DNAでは、アデニン(A)、チミン(T)、グアニン(G)、シトシン(C)の4種類の塩基が使われていますが、
RNAではチミンの代わりにウラシル(U)が使われ、A、U、G、Cの4種類となります。
補足:なぜDNAではチミン、RNAではウラシルを使うのか
なぜDNAではチミン(T)を使っているのに、RNAではウラシル(U)を使うのでしょうか?
RNAで、Uが使われるのは、Uの方がTよりも構造が単純で、合成にかかるエネルギーが少ないためです。RNAは一時的に作られてすぐに分解される「使い捨てのメモ」のような役割なので、エネルギーを節約して手早く作れる材料であるUが使われていると考えられます。
一方、DNAは体の設計図として長期間保存される必要があるため、情報をできるだけ正確に保つ必要があります。
ここで重要なのが「シトシン(C)が自然にウラシル(U)に変わってしまう」という現象です。
これは、細胞内で起こる酸化的脱アミノ反応によって、シトシンからアミノ基(−NH₂)が外れて代わりに酸素原子が付加されることで起こります。
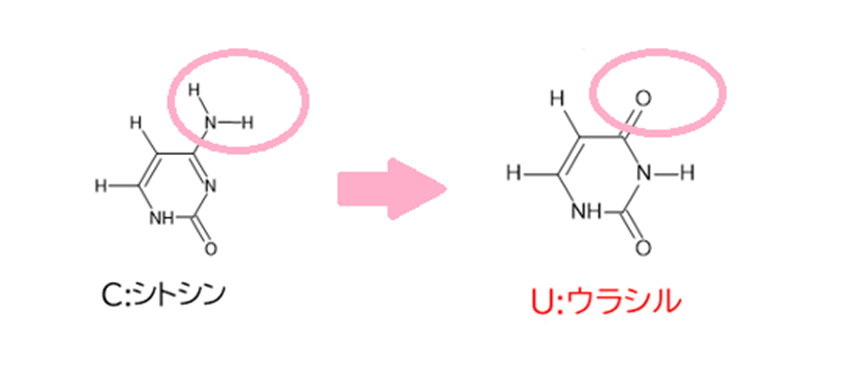
ここで問題になるのが、DNAがもし元からUを使っていたら、どのUが「壊れたC」なのか分からなくなる、ということです。
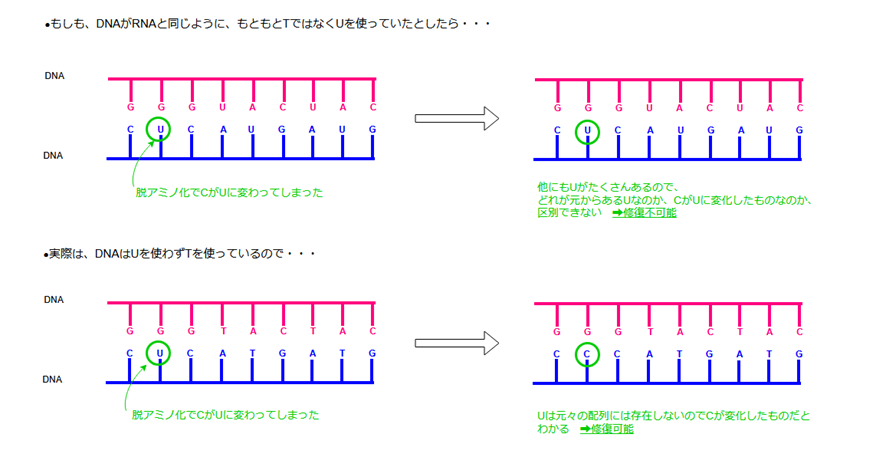
上の図の上段は、もしDNAがもともとTではなくUを使っていたとしたら、という例です。
修復すべきUの他に、もともとの塩基配列に含まれているUが存在することになるため、修復酵素はどのUが「Cが変化してできたもの」なのか判断できません。すべてのUが正しいものに見えてしまうのです。
一方、下は実際のDNAで、もともとUは使わずTを使っている例です。
この場合は、もしUが1つでも見つかれば、それは明らかに「Cが壊れてUになってしまったもの」だとわかります。したがって、UをCに修復することができるのです。
こうした理由から、DNAは情報を守るためにTを使い、RNAは効率よく使い捨てできるようにUを使っているのです。
転写のしくみ
mRNAは、DNAの情報を読み取ることで合成されます。
mRNAを合成するためには、DNAの特定の位置を読み取って、そこに対応するRNAを一塩基ずつつくる精密なしくみが必要です。
ここからは、どのようにしてDNAからmRNAが転写されるのか、そのしくみを見ていきましょう。
原核生物と真核生物とでは転写の仕方に違いがあるので、以下ではそれぞれの仕組みを説明します。
原核生物の転写
まずは転写の起点となる場所を決める必要があります。
DNAの配列のうち、転写の起点となる配列が含まれる領域をプロモータ領域と呼びます。
原核生物の場合は、プロモータ領域内に、-35領域と-10領域と呼ばれる部分があります。
-35と-10という数字は、RNAとして転写される最初の塩基に対応するDNAの位置を「+1」とし、それよりもそれぞれ35塩基分、10塩基分手前にある位置を意味しています。
原核生物の場合はRNAポリメラーゼ単体で転写が行われ、RNAポリメラーゼを構成するσ因子と呼ばれるサブユニットがこのプロモータと特異的に結合します。
-35領域と-10領域の2箇所を認識することで、RNAポリメラーゼが2本のDNA鎖のどちらに沿って進んでいくか向きを正しく決定することができます。
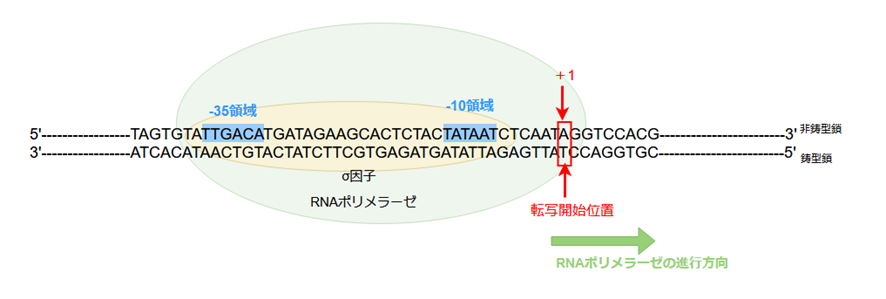
転写が開始されるとσ因子はRNAポリメラーゼから離れ、転写が進んでいきます。
転写の終点にあたるターミネータと呼ばれる特定の配列を転写すると転写が完了します。
すると遊離していたσ因子が再びRNAポリメラーゼに結合し、DNAの別の箇所に移動して次の転写に進みます。
真核生物の転写
真核生物の場合は、原核生物のように1種類のRNAポリメラーゼだけで転写を行うことはできず、転写基本因子と呼ばれる複数のタンパク質の助けを必要とします。
RNAポリメラーゼにもRNAの種類に応じていくつか種類がありますが、mRNAの転写にかかわるのはRNAポリメラーゼⅡと呼ばれるRNAポリメラーゼです。
真核生物の場合にはTATAボックスと呼ばれる、チミン(T)とアデニン(A)が繰り返された特定の配列があり、この部分がプロモータ領域となります。
まず、TFIIDという転写基本因子が、TBP(TATAボックス結合タンパク)と呼ばれる部分でこのTATAボックスを認識して結合します。
TBPがTATAボックスに結合することでDNAが曲がり、TFIIBという転写基本因子が結合できる構造が生まれます。
これらを目印にして、TFIIFと結合したRNAポリメラーゼⅡ、TFIIE、TFIIHが集まってきます。
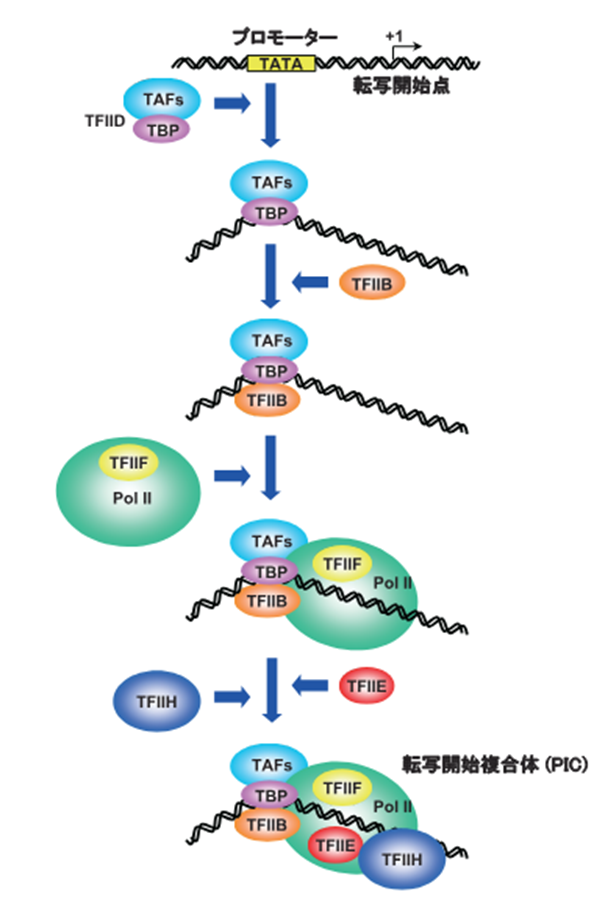
TFIIHはATP加水分解のエネルギーを利用して転写開始点付近のDNAの二重らせんをほどきます。
またRNAポリメラーゼⅡにはC末端ドメインと呼ばれる長く突き出した尾部があり、TFIIHによってこの尾部がリン酸化されると転写が開始します。
また、前回記事で説明したように、真核生物はDNAが高度に折りたたまれたクロマチン構造を持つため、遠くの塩基配列であっても比較的空間的に近くに位置する場合があります。
DNAの中にはTATAボックスよりも数千塩基離れた場所にエンハンサーと呼ばれる配列があり、ここにアクチベーターというタンパク質が結合し、さらにそのアクチベーターが基本転写因子に結合することで転写が促進されます。

転写後修飾
ここまで、DNAからmRNAへの転写の基本的な流れを見てきました。
次に、真核生物に特有の転写後修飾について説明します。
原核生物は核を持たないため、DNAとタンパク質を合成する場が同じ細胞質ですが、
真核生物の場合は核内でDNAの転写を行った後、RNAを細胞質に移動してからタンパク質に翻訳する必要があります。
真核生物の場合、DNAから転写された直後のmRNA(pre-mRNA)は、細胞質に移動する前に転写後修飾を受けて、初めて成熟したmRNAになります。
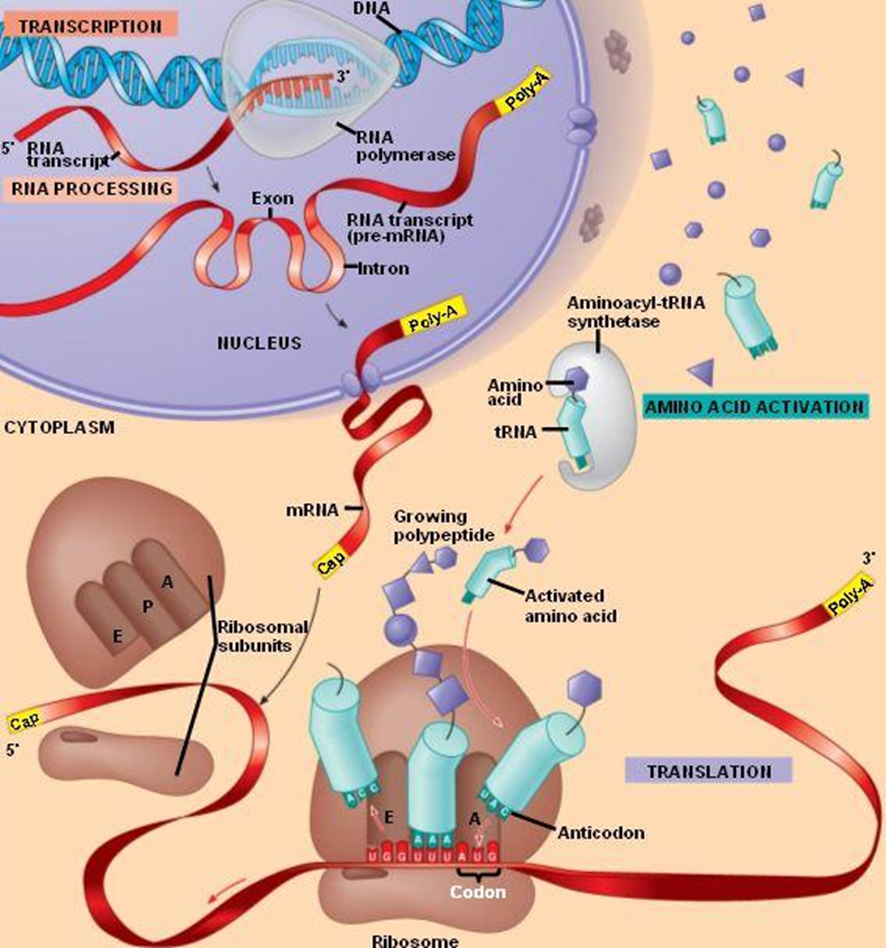
転写後修飾には、
- 5’キャップ付加
- スプライシング
- ポリアデニル化
という3つの工程があります。それぞれ見ていきましょう。
5’キャップ付加
5’キャップ付加は、pre-mRNAの5’末端への修飾です。
pre-mRNAはRNAポリメラーゼにより5’→3’の方向で合成されていますので、5’キャップは転写が完了するはるか前に優先して行われます。
グアノシン(塩基:グアニンと糖:リボースが結合した化合物)にメチル基が結合してできた、7-メチルグアノシンという物質がpre-mRNAの5’末端に、下図のような特殊な形で結合します。
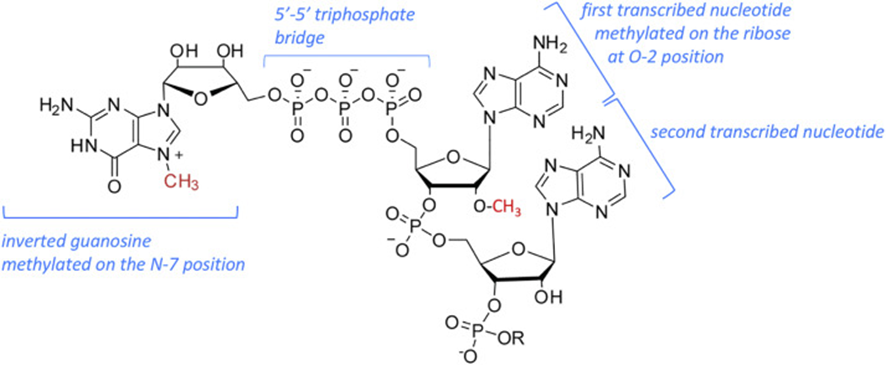
ポリアデニル化(ポリA付加)
ポリアデニル化(ポリA付加)は、転写完了直後に行われるpre-mRNAの3’末端への修飾です。
3’末端付近にはポリAシグナルとよばれる6塩基配列(AAUAAAなど)が存在します。
その配列の10~30塩基下流でmRNAが切断され、切断された部分に数百ヌクレオチド程度のアデニン(A)が連なった鎖(ポリA尾部)が付加されます。
これを「ポリアデニル化」または「ポリA付加」と呼びます。
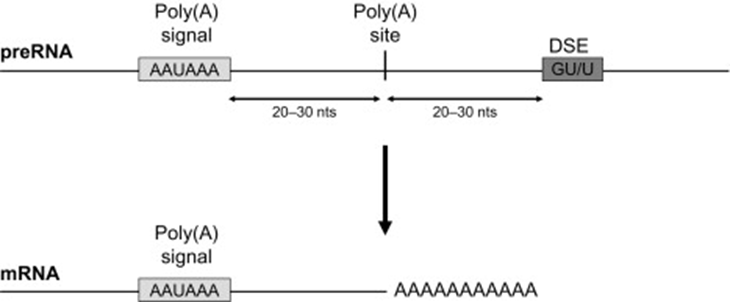
ポリA付加は次に説明するスプライシングと同時進行またはその後に行われます。
5’キャップ付加とポリアデニル化による両末端への修飾は、核から細胞質への移動に際してmRNAを安定化させ、翻訳の起点と終点の目印などの役割を果たします。
スプライシング
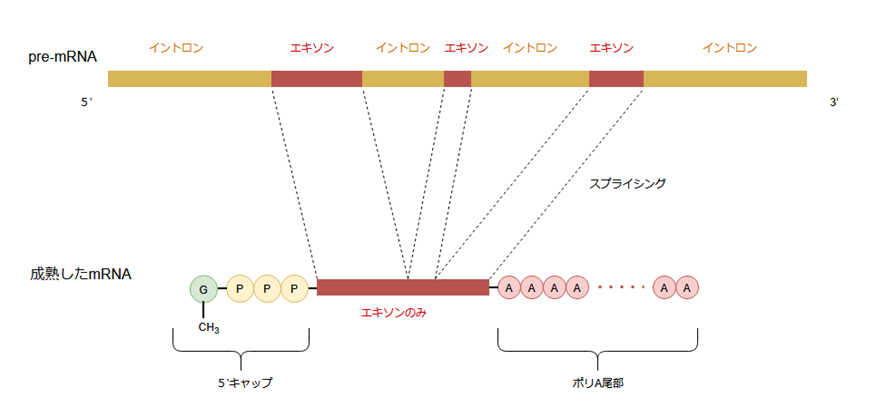
真核生物のDNAには、エキソンと呼ばれる合成に必要な情報を含む配列と、イントロンと呼ばれるタンパク質に翻訳されない多くの不要な配列が含まれています。
転写においてはこのイントロンの部分もそのまま転写されるため、翻訳前にpre-mRNAに含まれるイントロンを除去してエキソンだけをつなげ直す必要があります。
この除去プロセスは、スプライシングと呼ばれます。
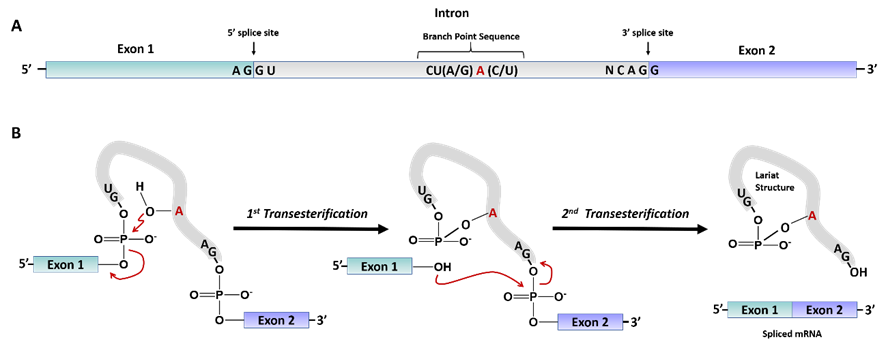
スプライシング時は、まずイントロンに含まれる分枝点と呼ばれる部分のアデニンが、5’側のエキソンとの結合部のリン酸基を攻撃して5’側のエキソンとイントロンを切断します。
次に、遊離したエキソンの3’側のOH基がイントロンの3’側のエキソンとの結合部のリン酸基を攻撃して切断します。
これによりエキソン同士が結合し、間に入っていたイントロンはラリアット(投げ縄形)になって外れます。こうして除去されたイントロンは核内で分解されます。
補足:イントロンがあるのは何のため?
ところで、真核生物にイントロンが含まれているのは、単なる無駄ではなく、生物にとって大きなメリットがあると考えられています。
イントロンの存在により、「スプライシングの仕方を変える」ことができるようになり、同じpre-mRNAから異なるmRNAを作ることができます。
これを選択的スプライシングと呼びます。
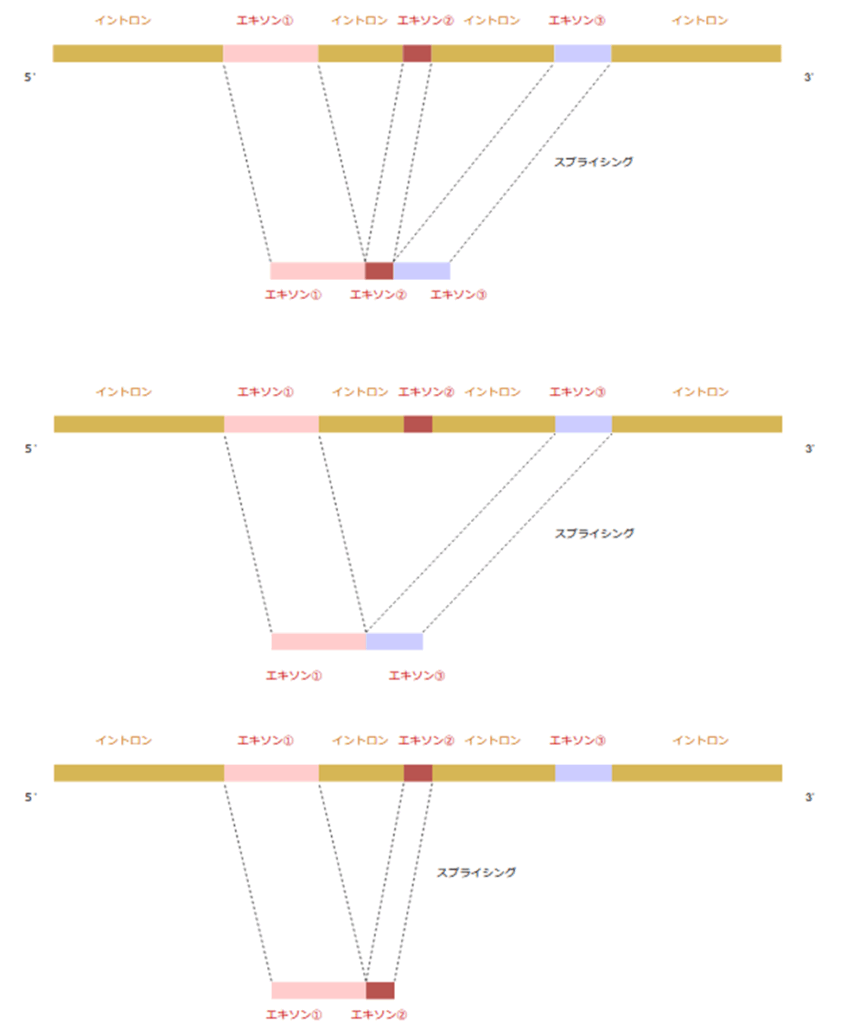
結果として、1つの遺伝子から複数のタンパク質を作り出すことができるようになり、生物のタンパク質の多様性が高まります。これは進化上の大きな利点です。
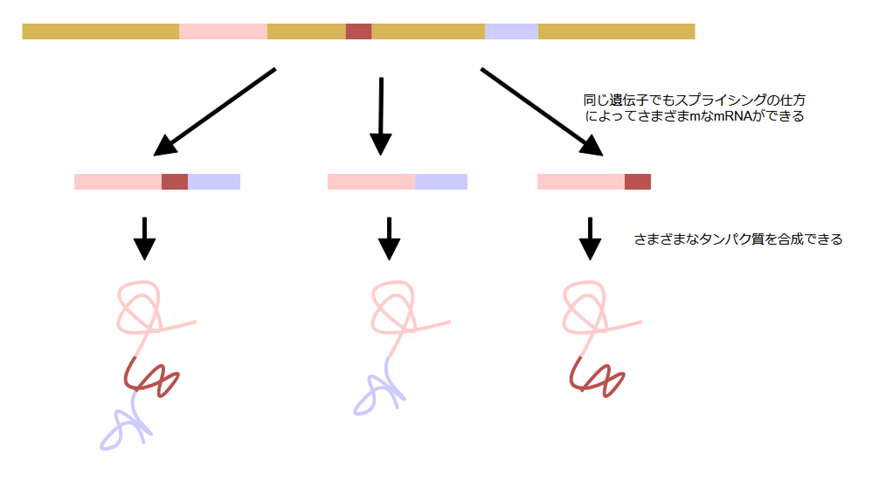
一方、原核生物の遺伝子には基本的にイントロンは存在せず、DNA配列のほとんどがそのままタンパク質合成に使われます。
また、原核生物と真核生物ではこのようなイントロンの有無による違いがあるために、真核生物のDNAを原核生物の体内で発現させる場合には、少し話が複雑になります。
前回記事ではグルコースセンサで使用する酵素について、「目的の遺伝子(DNA)をPCRで増やして、それを使ってタンパク質をつくる」とざっくり説明しましたが、実はそれだけでは不十分な場合があります。
たとえば、一部のグルコースセンサで使用される真核生物のカビから得られるFAD型グルコースデヒドロゲナーゼ遺伝子にはイントロンが含まれており、そのままでは大腸菌で発現できません。こうした場合は、mRNAからイントロンを除いたcDNAというものを合成して用いる方法がとられます(この点については次回詳述します)。
翻訳のしくみとリボソームのはたらき
mRNAに写された情報は、リボソームでアミノ酸の配列へと読み替えられます。
これが翻訳と呼ばれるプロセスです。
真核生物では、リボソームが5’キャップ構造を認識してスキャンを始め、最初に出会ったAUG(開始コドン)から翻訳を開始します。
原核生物では、mRNA上のシャイン・ダルガーノ配列と呼ばれる配列が目印となり、その近くのAUGから翻訳が始まります。
また、両者には転写と翻訳のタイミングにも大きな違いがあります。
真核生物では、前述のとおり、転写は核内で、翻訳は細胞質で行われるため、転写が終わってから翻訳が始まります。
一方、原核生物では、核を持たないため、転写も翻訳も同じ細胞質で行われます。
したがって、mRNAがまだ転写されている途中でもそのmRNAにリボソームが結合し、翻訳が同時進行で始まるのが基本です。これを共翻訳と呼びます。
このように、翻訳が「いつ」「どこで」始まるかには違いがありますが、いずれの場合も、mRNAをもとにアミノ酸をつなげてタンパク質をつくるという翻訳の基本的な流れは共通です。
ここからは、翻訳のしくみそのものに注目して見ていきましょう。
コドン
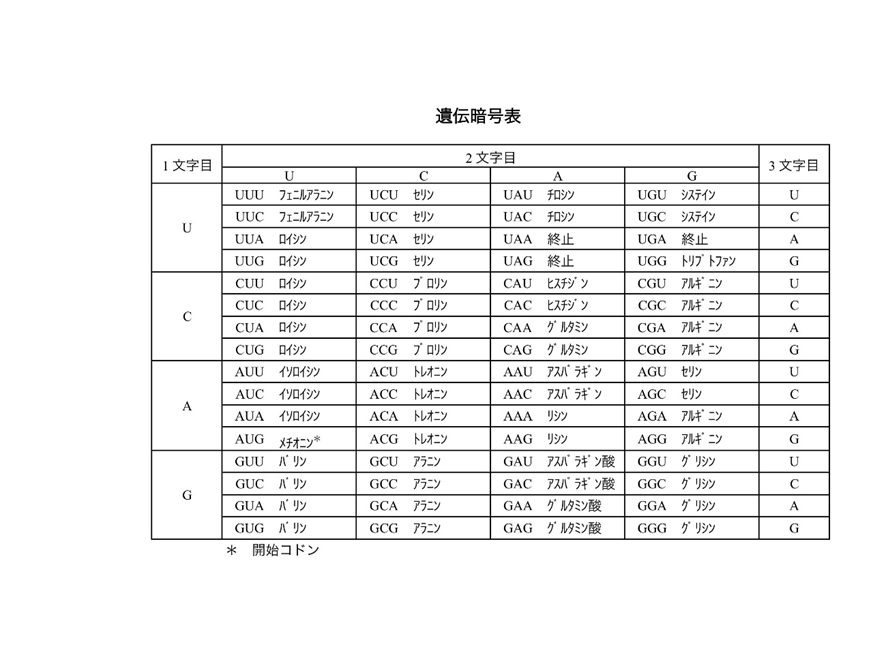
mRNAに転写された塩基は3つで1組のコドンという単位で構成されており、1つ1つのコドンは、翻訳の開始点、特定のアミノ酸、翻訳の終点を表しています。
例えば、CAAはグルタミン、UUAはロイシン、CGAはアルギニン、というアミノ酸を指定します。
1つのコドンは1種類のアミノ酸しか指定しませんが、1種類のアミノ酸に対しては複数のコドンが対応することがあり、これを遺伝暗号の冗長性と呼びます。
この冗長性は、変異が起きた際にタンパク質の性質に影響を与えにくくする働きがあると考えられています。
tRNA
コドンに対応するアミノ酸を運んでくるのがtRNA(transfer RNA)という分子です。
例えるなら、レシピメモ(mRNA)を見ながら、必要な食材(アミノ酸)を順番に運んでくる役割を担います。
tRNAはクローバーのような形をしていて、先端にアミノ酸、中央にmRNAのコドンと相補的なアンチコドンを持っています。
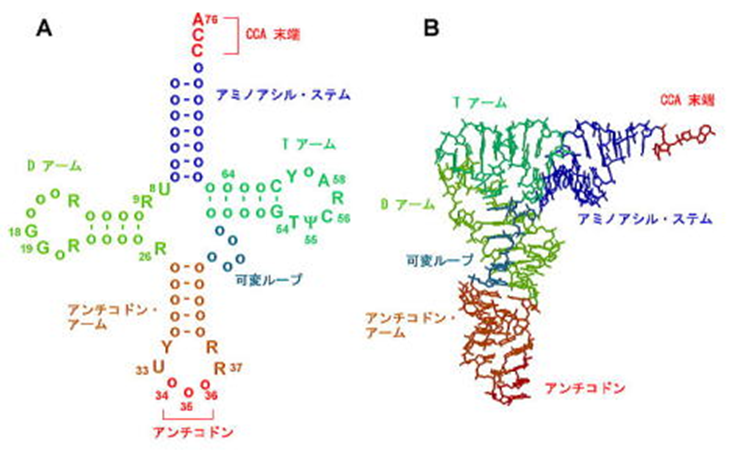
3次元の立体構造は上図の右側のようにL字型をしています。

ちょうどクローバーの左右の二枚の葉が内側に折り込まれてL字型の構造を安定させるようになっています。
リボソーム内での翻訳プロセス
翻訳は細胞質中のリボソームという物質の中で行われます。
まず真核生物ではリボソームの小サブユニットが5’キャップを目印にmRNAの翻訳開始部位に結合します。原核生物ではリボソームがシャイン・ダルガーノ配列と呼ばれる特定の配列を目印に翻訳を開始します。
続いてtRNAがリボソームの小サブユニットに結合した開始コドン(AUG)を読んで、対応するアミノ酸(メチオニン)を運んできます。
メチオニンを運んできた開始tRNAがリボソームの小サブユニットに結合すると、それを基点に大サブユニットが合体します。
このとき、リボソーム内にPサイト・Aサイト・Eサイトと呼ばれる3つのサイトが形成されます。
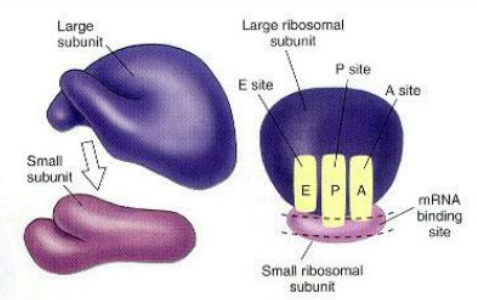
- Aサイト(Aminoacyl site)
mRNAのコドンに対応するアミノ酸を運んできたtRNAが新たに収まる場所です。 - Pサイト(Peptidyl site)
1つ前のtRNAが収まっている場所で、ここに入っているtRNAがペプチド鎖を持ちます。
最初は開始tRNAが収まっています。 - Eサイト(Exit site)
出口の役割を果たし、役目を終えて出ていくtRNAが一時的に収まる場所です。
先ほどの料理の例で言うなら、Aサイトは食材の受け取り口、Pサイトは調理場、Eサイトは使い終わった道具の返却口といったところでしょうか。
Aサイトに新たなtRNAが入ってくると、このtRNAが運んできたアミノ酸と、Pサイトに入っていたtRNAのアミノ酸がペプチド結合します。
AサイトのtRNAは、PサイトのtRNAが持っていたペプチド鎖に自分が新たに持ってきたアミノ酸1個を足したペプチド鎖を持つようになります。
役目を終えたPサイトのtRNAはEサイトに移動してリボソームから出ていきます。
Aサイトに入ってきたtRNAがPサイトに移動し、再びAサイトが空席になります。
そしてまたAサイトに新たなtRNAが入ってきて、同じプロセスが繰り返され、ペプチド鎖がどんどん伸長していきます。
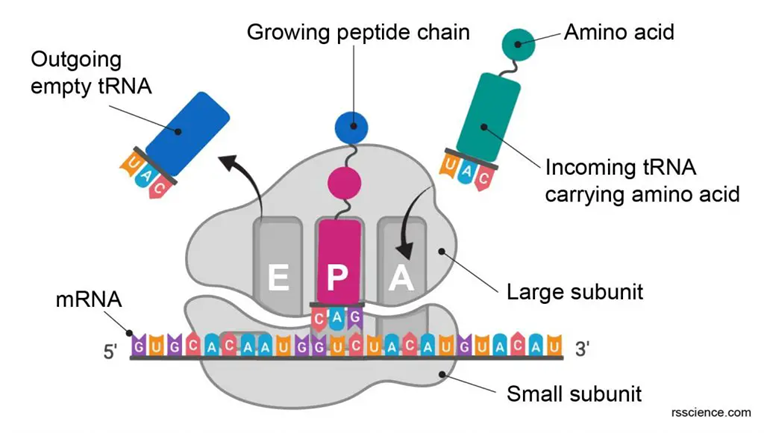
そして終了を意味するコドン(終止コドン)に到達すると、翻訳は終了します。
この終止コドンは、UAA、UAG、UGAの3種類があり、どれも「ここでタンパク質の合成を終わらせる」という合図を表しています。
終止コドンは、どのtRNAのアンチコドンにも対応していないため、そこに新たなtRNAがアミノ酸を運んで来ることはありません。そのかわり、終結因子と呼ばれる特殊なタンパク質がリボソームに結合します。
この終結因子の働きで、リボソームはmRNAとtRNAから切り離され、完成したタンパク質が細胞内に放たれます。
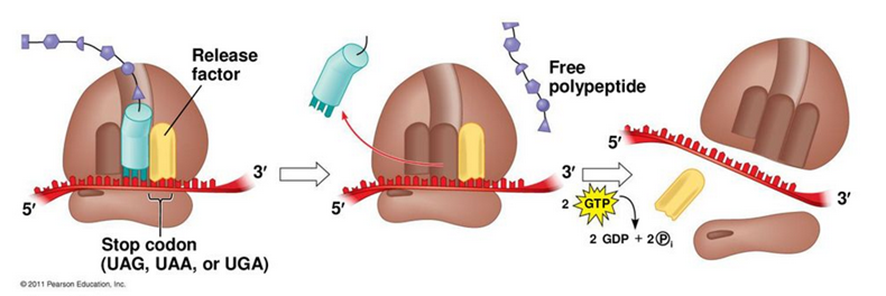
こうして、tRNAが運んでくるアミノ酸の並びによって、最終的に目的のタンパク質が合成されます。
原核と真核における違いのまとめ
ここまで、転写や翻訳の各段階で原核生物と真核生物の違いに触れてきました。
最後にこれらの違いを表にまとめると以下のようになります。
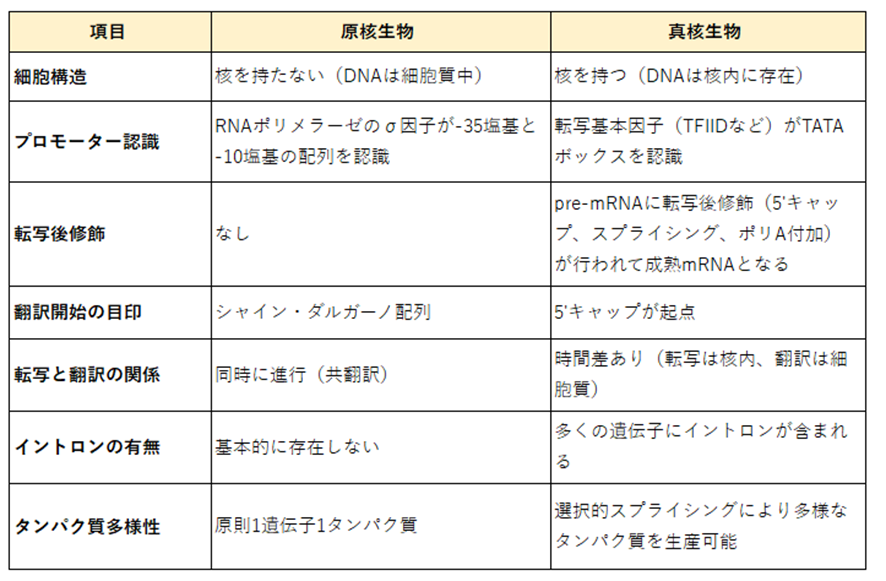
[参考]
・Essential細胞生物学
・「転写とRNAポリメラーゼ」国立遺伝学研究所マルチメディア資料館
・「翻訳とリボソーム」国立遺伝学研究所マルチメディア資料館








コメントを残す